آموزش شبکه های عصبی بازگشتی (Recurrent neural network) بخش سوم : Backpropagation
بسم الله الرحمن الرحیم
در بخش قبل(بخش دوم) با هم چگونگی پیاده سازی فاز پیشرو (forward pass) در یک شبکه عصبی بازگشتی (Recurrent Neural network) را به انجام رساندیم. در این پست به فاز دوم پیاده سازی یعنی پیاده سازی عملیات پس انتشار (Backpropagation) در یک شبکه عصبی بازگشتی خواهیم پرداخت.
در این بخش با مفاهیم “پس انتشار در زمان” و “پس انتشار کوتاه” شده نیز آشنا خواهیم شد.
حالا نوبت به پیاده سازی بخش پس انتشار است. اما قبل از انکه ادامه دهیم اجازه دهید با هم مروری بر این فرآیند داشته باشیم و ببینیم چگونه باید به پیش برویم .
عملیاتهای صورت گرفته در بخش forward مشخص هستند اما برای اینکه قادر باشیم این فاز را بدرستی پیاده سازی کنیم نیاز به جزییات بیشتری داریم . میدانیم که در یک شبکه RNN ما بطور خاص با یک حلقه سرو کار داریم به همین جهت گرادیان هایی که به ازای عملیات انجام شده بدست می آوریم را بایستی با هم جمع کنیم تا در نهایت بدرستی تاثیرات صورت گرفته در فاز forward را لحاظ کرده باشیم .
همانطور که بالا دیدیم فاز forward ما متشکل از ۲ عملیات کلی بود که به قرار زیراند :
برای انجام پس انتشار ما از گام زمانی اخر شروع کرده خطا را محاسبه میکنیم و سپس در یک حلقه محاسبات را به ازای تمامی گام های زمانی ادامه میدهیم .
فرض ما در اینجا این است که شما با عملیات پس انتشار(backpropagation) آشنایید بنابر این در اینجا با جزییات کامل پس انتشار(که پس انتشار چیست) آشنا نمیشویم انشاءالله در مورد عملیات پس انتشار و مبحث گرادیان ها در مطلبی جداگانه بطور مفصل صحبت خواهم کرد اما بطور کوتاه باید بدانیم که الگوریتم پس انتشار (خطا) یک الگوریتم credit assignment است. یعنی کار این الگوریتم منتسب کردن پاداش یا جزای هر پارامتر نسبت به نتیجه بدست امده نهایی است. اینکه هر پارامتر چقدر در نتیجه بدست امده دخیل بوده و در صورت نتیجه صحیح و یا غلط به چه میزان بایستی تغییر کند. این تمام چیزی است که این الگوریتم مسئولیت انجام آن را دارد. ستون فقرات این الگوریتم را هم گرادیان ها تشکیل میدهند. کاری که ما در اینجا بایستی انجام دهیم مشخص کردن میزان تاثیر هرکدام از متغییرها بر روی نتیجه بدست آمده نهایی است. برای اینکار ما از مشتق جزئی استفاده میکنیم همانطور که حتما میدانید مشتق جزئی در مورد توابع چند متغیره، به مشتق تابع نسبت به یکی از متغیرها با ثابت نگهداشتن سایر متغیرها گفته میشود. بعنوان مثال در عبارت مشتق جزئی
نسبت به
برابر با
خواهد بود. و اگر از تابع
نسبت به
بخواهیم مشتق بگیریم نتیجه برابر با
خواهد بود. زمانی که با مشتق جزئی سرو کار داریم از نماد
استفاده میکنیم. برای یادآوری این مبحث میتوانید به این نوشته کوتاه و بسیار خوب مراجعه کنید.
خب به بحث اصلی برگردیم. برای محاسبه گرادیان ها ما بصورت زیر بایستی عمل کنیم :
- محاسبه خطا :
- محاسبه گرادیان عبارت خروجی :
- محاسبه گرادیان عبارت مربوط به حالت مخفی جدید :
ابتدا سعی میکنیم خطا را محاسبه کرده و سپس مشتق جزئی برای رابطه را بدست بیاوریم .
برای محاسبه مشتق عبارت فوق بهتر است از فرم ساده شده آن استفاده کنیم تا فرآیند مشتق گیری آسان تر باشد. ما میتوانیم تابع فوق را بصورت در نظر بگیریم. بعد از اینکار مشتق ما بصورت زیر خواهد بود :
از انجایی که در این بخش ما بصورت مستقیم با خطا در ارتباط هستیم در نتیجه خطا را بصورت مستقیم میتوانیم لحاظ کنیم بنابر این خواهیم داشت :
پیاده سازی این عبارات در پایتون بصورت زیر خواهد بود :
|
1 2 3 4 5 6 7 8 |
# must have the shape of W3 which is (output_dim_size, hidden_state_size) dW3 = np.dot(e.T, h) dh = np.dot(e, W3) # dbo =e.1, since we have batch we use np.sum # e is a vector, when it is subtracted from label, the result will be added to dbo dbo = np.sum(e, axis=0) |
نکته:
در تعاریف بالا یک مشکل وجود دارد و آن بحث لحاظ کردن حالات مخفی و تاثیرات آنها بر یکدیگر است . همانطور که در فاز forward حالت قبلی در گام زمانی بعدی مورد استفاده قرار میگرفت به همین صورت نیز مشتق حالت مخفی از گام زمانی جدیدتر به گام زمانی قدیمی تر بایستی منتقل شود. از آنجایی که در فاز پس انتشار ما از اخرین گام زمانی شروع کرده و بعد از این گام زمانی دیگری وجود ندارد در نتیجه در شروع کار = ۰ خواهد بود. اما این مقدار هر بار بروز شده و در گام زمانی قبل تر مورد استفاده قرار میگیرد.
همانطور که در تصویر زیر میبینیم هر سلول فعلی را به گام زمانی بعدی ارسال میکند. به همین صورت در زمان بک پراپگیشن هم
علاوه بر محاسبه گرادیان مبتنی بر خروجی ، گرادیان حاصل از گام زمانی بعدی نیز بایستی لحاظ شود.
در تصویر زیر این مهم مشخص شده است, نکته قابل اهمیت dh است به همین منظور تنها جهت جریان forward و همینطور گرادیان متناظر با آن مشخص شده است .
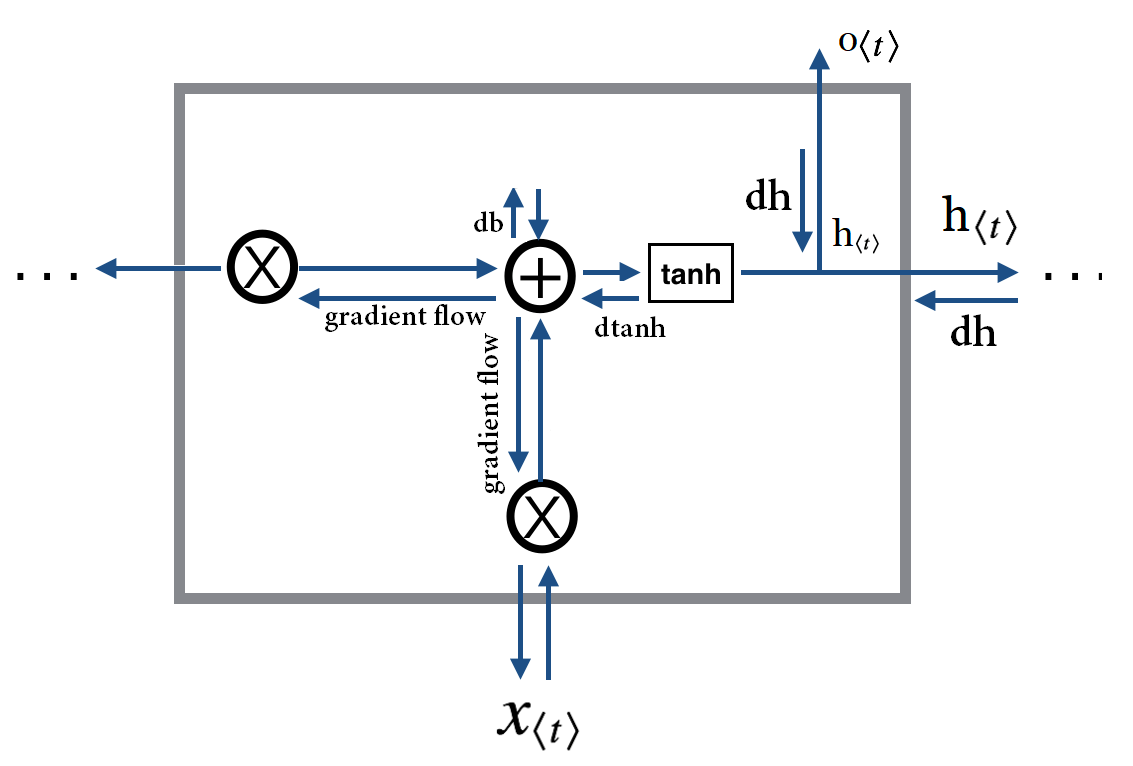
به همین خاطرمحاسبه بصورت زیر خواهد بود :
*** QuickLaTeX cannot compile formula:
dh = W_3.error + dh_{from\_next\_timestep}}
*** Error message:
Extra }, or forgotten $.
leading text: $dh = W_3.error + dh_{from\_next\_timestep}}
و از انجایی که شروع عملیات پس انتشار از گام زمانی اخر است و گام زمانی بعدی وجود ندارد مقدار در ابتدا صفر میباشد.
به همین صورت پیاده سازی عبارات فوق در زبان پایتون بصورت زیر خواهند بود :
|
1 2 3 4 5 6 7 8 9 10 |
# must have the shape of W3 which is (output_dim_size, hidden_state_size) dW3 = np.dot(e.T, h) # when calculating the dh, we also add the dh from the next timestep as well # when we are in the last timestep, the h_gradient_or_dh is initially zero. dh = np.dot(e, W3) + h_gradient_or_dh # dbo =e.1, since we have batch we use np.sum # e is a vector, when it is subtracted from label, the result will be added to dbo dbo = np.sum(e, axis=0) |
حالا نیازمند محاسبه مشتقات جزئی برای عبارت دوم هستیم . همانطور که تازه دیدیم میتوانیم این عبارت را نیز بصورت ساده سازی کنیم. از انجایی که تابع
ما در اینجا در اصل
است و مشتق
برابر با
*** QuickLaTeX cannot compile formula: 1-tan(u)^2\partialu *** Error message: Undefined control sequence \partialu. leading text: $1-tan(u)^2\partialu
است. بنابر این خواهیم داشت و با جایگزین کردن خواهیم داشت:
چرا که
*** QuickLaTeX cannot compile formula: dtanh = (1-tanh(u)^2\partialu) *** Error message: Undefined control sequence \partialu. leading text: $dtanh = (1-tanh(u)^2\partialu
پس
به همین صورت مشتق سایر عبارتها را بصورت زیر بدست می آوریم :
حالا اگر بخواهیم معادل این دستورات را در پایتون داشته باشیم بصورت زیر عمل میکنیم.
توجه :
دقت کنید که ما در اینجا ضرب بین بردارها و ماتریس ها را داریم و ابعاد مشتقات برابر با اندازه بردار یا ماتریس متناظر آن است(همانطور که قبلا گفتیم در مشتق ما بدنبال میزان تاثیر هر “پارامتر” بر روی نتیجه نهایی هستیم. پس به ازای هر پارامتر قابل یادگیری (هر دارایه ماتریس یا بردار) ما یک مقدار خواهیم داشت. همینطور توجه کنید که ما تنها به پارامترهای قابل ترین اهمیت میدهیم.(کد زیر را ببینید)
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
# the input part dtanh = (1 - h * h) * dh # compute the gradient of the loss with respect to W1 # this is actually not needed!, because its not a tunable parameter! # dxt = np.dot(dtanh, W1.T) # must have the shape of (input_dim_size, hidden_state_size) dW1 = np.dot(xt.T, dtanh) # compute the gradient with respect to W2 dh_prev = np.dot(dtanh, W2.T) # shape must be (HiddenSize, HiddenSize) dW2 = np.dot(h_prev.T, dtanh) # dbh += dtanh.1, we use sum, since we have a batch dbh = np.sum(dtanh, axis=0) |
بعد از مشخص کردن موارد بالا پیاده سازی کامل این فاز براحتی قابل انجام خواهد بود. دو تابع زیر وظیفه پیاده سازی موارد مطرحی بالا را بعهده دارند :
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 |
def rnn_cell_backward(xt, h, h_prev, output, true_label, h_gradient_or_dh, W1, W2, W3 ): """ xt: is the input in the form of (batch_size, input_dim_size) h : is the next hidden state h_prev: is the previous hidden state output: is the output of the rnn cell! true_label: is the true label for the current output h_gradient_or_dh: The dh which in the beginning is zero and is updated as we go backward in the backprogagation. the dh for the next round, would come from the dh_prev. remember the backward pass is essentially a loop! and we start at the end and traverse back to the beginning! """ e = np.copy(output) # This is error_t = output_t - label_t e[np.arange(e.shape[0]), np.argmax(true_label, axis=1)] -=1 # This is used for our loss to see how well we are doing during training. per_ts_loss = output[np.arange(output.shape[0]), np.argmax(true_label, axis=1)].sum() # this is the non-vectorized version of the above snippet # per_ts_loss = 0 # for i, idx in enumerate(np.argmax(true_label, axis=1)): # e[i, idx] -=1 # per_ts_loss += output[i, idx] dW3 = np.dot(e.T, h) # when calculating the dh, we also add the dh from the next timestep as well # when we are in the last timestep, the h_gradient_or_dh is initially zero. dh = np.dot(e, W3) + h_gradient_or_dh # dbo = e.1, since we have batch we use np.sum # e is a vector, when it is subtracted from label, the result will be added to dbo dbo = np.sum(e, axis=0) # the input part dtanh = (1 - h * h) * dh # compute the gradient of the loss with respect to W1 # this is actually not needed! bcause Xt is not a tunable # parameter, thererfore we dont need it gradient. # dxt = np.dot(dtanh, W1.T) # must have the shape of (input_dim_size, hidden_state_size) dW1 = np.dot(xt.T, dtanh) # compute the gradient with respect to W2 dh_prev = np.dot(dtanh, W2.T) # shape must be (HiddenSize, HiddenSize) dW2 = np.dot(h_prev.T, dtanh) # dbh += dtanh.1, we use sum, since we have a batch dbh = np.sum(dtanh, axis=0) return dW1, dW2, dW3, dbh, dbo, dh_prev, per_ts_loss def rnn_layer_backward( Xt, labels, H, O, W1, W2, W3, bh, bo ): """ Xt: The input in the form of (batch_size, time-step, input_dim_size) labels: The labels for each output with the shape (batch_size, time-step, outputsize) H : All the hidden states from the forward pass with the shape (batch_size, time-step, hidden_state_size) O: All the outputs from the forward pass W1,W2,W3,bh and bo: Are network parameters. """ dW1 = np.zeros_like(W1) dW2 = np.zeros_like(W2) dW3 = np.zeros_like(W3) # must have the shape(batch, hiddensize) dbh = np.zeros_like(bh) # must have the shape(batch, outputsize) dbo = np.zeros_like(bo) dh = np.zeros_like(H[:, 0, :]) _, T_x , _= Xt.shape loss = 0 for t in reversed(range(T_x)): dw1, dw2, dw3, dbh, dbo, dh_prev, per_ts_loss = rnn_cell_backward(Xt[:, t, :], H[:, t, :], H[:, t - 1, :], O[:, t, :], labels[:, t, :], dh, W1, W2,W3 ) dh = dh_prev dW1 += dw1 dW2 += dw2 dW3 += dw3 dbh += dbh dbo += dbo # Update the loss by substracting the cross-entropy term of this time-step from it. loss -= np.log(per_ts_loss) return dW1, dW2, dW3, dbh, dbo, dh, loss |
به منظور تست توابع پیاده سازی شده نیز میتوان بصورت زیر عمل کرد :
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 |
import numpy as np np.random.seed(1) input_dim_size = 3 hidden_state_size = 5 outputsize = 3 batch = 2 timesteps = 4 W1 = np.random.randn(input_dim_size, hidden_state_size) W2 = np.random.randn(hidden_state_size, hidden_state_size) W3 = np.random.randn(outputsize, hidden_state_size) bh = np.random.randn(hidden_state_size) bo = np.random.randn(outputsize) Xt = np.random.rand(batch, timesteps, input_dim_size) # label has the same shape as input, because we are creating outputs just like the inputs. Labels = np.random.rand(batch, timesteps, input_dim_size) h_previous = np.zeros(shape=(batch, hidden_state_size)) Outputs, HiddenStates = rnn_layer_forward(Xt, hidden_state_size, W1, W2, W3,bh, bo) dW1, dW2, dW3, dbh, dbo, dh, loss = rnn_layer_backward(Xt, Labels, HiddenStates, Outputs,W1, W2, W3,bh, bo ) print(dW1.shape) print("dh_prev[0] =", dh[0]) print("dh.shape =", dh.shape) print("dW1[0] =", dW1[0]) print("dW1.shape =", dW1.shape) print("dW2[0] =", dW2[0]) print("dW2.shape =", dW2.shape) print("dbh[0] =", dbh[0]) print("dbh.shape =", dbh.shape) |
خروجی :
|
1 2 3 4 5 6 7 8 9 10 11 |
(3, 5) dh_prev[0] = [-0.44595092 -0.0868595 -0.77449515 -0.50100593 0.35927758] dh.shape = (2, 5) dW1[0] = [-1.96168867 0.86751548 5.07050684 0.13683888 0.24913199] dW1.shape = (3, 5) dW2[0] = [-0.70572562 -0.53948113 -0.28133053 1.30360588 0.15237789] dW2.shape = (5, 5) dbh[0] = -3.859417642155729 dbh.shape = (5,) |
پیاده سازی کامل :
در نهایت تمام پیاده سازی ما بصورت زیر خواهد بود :
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193 194 195 196 197 198 199 200 201 202 203 204 205 206 207 208 209 210 211 212 213 214 215 216 217 218 219 220 221 222 223 224 225 226 227 228 229 230 231 232 233 234 235 236 237 238 239 240 241 242 243 244 245 246 247 248 249 250 251 252 253 254 255 256 257 258 259 260 261 262 263 264 265 266 267 268 269 270 271 272 273 274 275 276 277 278 279 280 281 282 283 284 285 286 287 288 289 290 291 292 293 294 295 296 297 298 299 300 301 302 303 304 305 306 307 308 309 310 311 312 313 314 315 316 317 318 319 320 321 322 323 324 325 326 327 328 329 330 331 332 333 334 335 336 337 338 339 340 341 342 343 344 345 346 347 348 349 350 |
# in the name of Allah import numpy as np from sklearn.utils.extmath import softmax class RNNClass(object): def __init__(self, input_dim_size, outputsize, hidden_state_size=100, mode=1, clipping_threshold=0.5): np.random.seed(1) # weights and biases - primitive method! self.W1 = np.random.randn(input_dim_size, hidden_state_size) * 0.01 self.W2 = np.random.randn(hidden_state_size, hidden_state_size) * 0.01 self.W3 = np.random.randn(outputsize, hidden_state_size) * 0.01 # to fight vanishing gradient to some extend, we need proper weight initialization # Xavier-Glorot initialization # self.W1 = np.random.uniform( -np.sqrt(1./input_dim_size), np.sqrt(1./input_dim_size),(input_dim_size, hidden_state_size)) # self.W2 = np.random.uniform( -np.sqrt(1./hidden_state_size), np.sqrt(1./hidden_state_size),(hidden_state_size, hidden_state_size) ) # self.W3 = np.random.uniform( -np.sqrt(1./hidden_state_size), np.sqrt(1./hidden_state_size),(outputsize, hidden_state_size) ) # This can be zero, it can also be randomly initialized self.bh = np.ones(shape=(hidden_state_size)) self.bo = np.ones(shape=(outputsize)) self.input_dim_size = input_dim_size self.outputsize = outputsize self.hidden_state_size = hidden_state_size self.mode = mode self.clipping_threshold = clipping_threshold print('W1: ', self.W1.shape) print('W2: ', self.W2.shape) print('W3: ', self.W3.shape) print('bh: ', self.bh.shape) print('bo: ', self.bo.shape) def rnn_cell_foward(self, xt, h0): """ Run the forward pass for a single timestep of a vanilla RNN that uses a tanh activation function. The input data has dimension D(vocabsize in case we have nlp use), the hidden state has dimension H, and we use a minibatch size of N. Inputs: - x: Input data for this timestep, of shape (Batch_size, vocabsize_or_basically_input_dim_size). - h0: Hidden state from previous timestep, of shape (Batch_size, HiddenSize) Returns : - h_t: Next hidden state, of shape (Batch_size, HiddenSize) - output: output , of shape (Batch_size, outputsize) """ h_t = np.tanh(np.dot(xt, self.W1) + np.dot(h0, self.W2) + self.bh) o = np.dot(h_t, self.W3.T) + self.bo o_t = softmax(o) return o_t, h_t def rnn_layer_forward(self, Xt, H=None): """ Runs a forward pass on the input data Xt and optional hiddenstate H. if H is initialized, the initial hiddenstate (h0) will be initialized from the last hiddenstate using this optional argument. Inputs: - Xt: The input data of shape (Batch_size, time_steps, input_dim_size) - H(Optional): an array containing hiddenstates from previous example of shape(Batch_size, timesteps, HiddenStateSize) Returns : - O: The output for the current layer of shape (Batch_size, timesteps, outputsize) - H: The hiddenstates for the current layer of shape (Batch_size, timesteps, HiddenStateSize) """ batch, T_x, input_dim_size = Xt.shape H = np.zeros(shape=(batch, T_x, self.hidden_state_size)) O = np.zeros(shape=(batch, T_x, self.outputsize)) if (self.mode == 1): h_previous = np.zeros(shape=(batch, self.hidden_state_size)) else: h_previous = H[:, -1, :] for t in range(T_x): output, h_t = self.rnn_cell_foward(Xt[:, t, :], h_previous) H[:, t, :] = h_t O[:, t, :] = output # Our current/new hiddenstate will be the previous hiddenstate for the next round, h_previous = h_t return O, H def rnn_cell_backward(self, xt, h, h_prev, output, true_label, dh_next): """ Runs a single backward pass once. Inputs: - xt: The input data of shape (Batch_size, input_dim_size) - h: The next hidden state at timestep t(which come from the forward pass) - h_prev: The previous hidden state at timestep t-1 - output : The output at the current timestep - true_label: The label for the current timestep, used for calcuating loss - dh_next: The gradient of hiddent state h (dh) which in the beginning is zero and is updated as we go backward in the backprogagation. the dh for the next round, would come from the 'dh_prev' as we will see shortly! Just remember the backward pass is essentially a loop! and we start at the end and traverse back to the beginning! Returns : - dW1 : The gradient for W1 - dW2 : The gradient for W2 - dW3 : The gradient for W3 - dbh : The gradient for bh - dbo : The gradient for bo - dh_prev : The gradient for previous hiddenstate at timestep t-1. this will be used as the next dh for the next round of backpropagation. - per_ts_loss : The loss for current timestep. """ e = np.copy(output) # correct idx for each row(sample)! idxs = np.argmax(true_label, axis=1) # number of rows(samples) in our batch rows = np.arange(e.shape[0]) # This is the vectorized version of error_t = output_t - label_t or simply e = output[t] - 1 # where t refers to the index in which label is 1. e[rows, idxs] -= 1 # This is used for our loss to see how well we are doing during training. per_ts_loss = output[rows, idxs].sum() # must have shape of W3 which is (vocabsize_or_output_dim_size, hidden_state_size) dW3 = np.dot(e.T, h) # dbo = e.1, since we have batch we use np.sum # e is a vector, when it is subtracted from label, the result will be added to dbo dbo = np.sum(e, axis=0) # when calculating the dh, we also add the dh from the next timestep as well # when we are in the last timestep, the dh_next is initially zero. dh = np.dot(e, self.W3) + dh_next # from later cell # the input part dtanh = (1 - h * h) * dh # dbh = dtanh.1, we use sum, since we have a batch dbh = np.sum(dtanh, axis=0) # compute the gradient of the loss with respect to W1 # this is actually not needed! we only care about tunable # parameters, so we are only after, W1,W2,W3, db and do # dxt = np.dot(dtanh, W1.T) # must have the shape of (vocab_size, hidden_state_size) dW1 = np.dot(xt.T, dtanh) # shape must be (HiddenSize, HiddenSize) dW2 = np.dot(h_prev.T, dtanh) # compute the gradient with respect to W2 dh_prev = np.dot(dtanh, self.W2.T) return dW1, dW2, dW3, dbh, dbo, dh_prev, per_ts_loss def rnn_layer_backward(self, Xt, labels, H, O): """ Runs a full backward pass on the given data. and returns the gradients. Inputs: - Xt: The input data of shape (Batch_size, timesteps, input_dim_size) - labels: The labels for the input data - H: The hiddenstates for the current layer prodced in the foward pass of shape (Batch_size, timesteps, HiddenStateSize) - O: The output for the current layer of shape (Batch_size, timesteps, outputsize) Returns : - dW1: The gradient for W1 - dW2: The gradient for W2 - dW3: The gradient for W3 - dbh: The gradient for bh - dbo: The gradient for bo - dh: The gradient for the hidden state at timestep t - loss: The current loss """ dW1 = np.zeros_like(self.W1) dW2 = np.zeros_like(self.W2) dW3 = np.zeros_like(self.W3) dbh = np.zeros_like(self.bh) dbo = np.zeros_like(self.bo) dh_next = np.zeros_like(H[:, 0, :]) hprev = None batchsz, T_x, inptdm = Xt.shape loss = 0 for t in reversed(range(T_x)): # this if-else block can be removed! and for hprev, we can simply # use H[:,t -1, : ] instead, but I also add this in case it makes a # a difference! so far I have not seen any difference though! if t > 0: hprev = H[:, t - 1, :] else: hprev = np.zeros_like(H[:, 0, :]) dw_1, dw_2, dw_3, db_h, db_o, dh_prev, e = self.rnn_cell_backward(Xt[:, t, :], H[:, t, :], hprev, O[:, t, :], labels[:, t, :], dh_next) dh_next = dh_prev dW1 += dw_1 dW2 += dw_2 dW3 += dw_3 dbh += db_h[0] dbo += db_o # Update the loss by substracting the cross-entropy term of this time-step from it. loss -= np.log(e) return dW1, dW2, dW3, dbh, dbo, dh_next, loss def update_parameters(self, dW1, dW2, dW3, dbh, dbo, lr): """ Updates the parameters. Inputs: - lr : the learning rate used for updaing the parameters. - dW1: The gradient for W1 - dW2: The gradient for W2 - dW3: The gradient for W3 - dbh: The gradient for bh - dbo: The gradient for bo Returns : - W1: The updated W1 - W2: The updated W2 - W3: The updated W3 - bh: The updated bh - bo: The updated bo """ self.W1 += -lr * dW1 self.W2 += -lr * dW2 self.W3 += -lr * dW3 self.bh += -lr * dbh self.bo += -lr * dbo return self.W1, self.W2, self.W3, self.bh, self.bo def clip(self, dW1, dW2, dW3, dbh, dbo, maxValue=0.5): ''' Clips the gradients' values between minimum and maximum. Arguments: The gradients "dW1", "dW2", "dW3", "db", "dby" maxValue -- everything above this number is set to this number, and everything less than -maxValue is set to -maxValue Returns: gradients -- "dW1", "dW2", "dW3", "db", "dby" ''' for gradient in [dW1, dW2, dW3, dbh, dbo]: np.clip(gradient, a_min=-maxValue, a_max=maxValue, out=gradient) return dW1, dW2, dW3, dbh, dbo def optimize(self, X, Y, H, learning_rate=0.01): """ Runs a complete forward-backward pass and then updates The weights. Inputs: - X: The input data of shape (Batch_size, time_steps, input_dim_size) - Y: The labels for the input data of shape (Batch_size, time_steps, outputsize) - H: (Optional), The hiddenstates of shape (Batchsize, timesteps, hiddenstateSize) that you can provide, so that in the forward pass, the initial hiddenstate uses its last timestep hiddenstate for the previous examples forward pass. Initially array must be all zeros, then after the first fowardpass, you return the resulting H, and for the next round, use that H! We will see how this works when we try to use our class on a test data! Returns : - loss - dW1: The gradient for W1 - dW2: The gradient for W2 - dW3: The gradient for W3 - dbh: The gradient for bh - dbo: The gradient for bo - H: The hiddenstates for this round of forward pas """ # Forward propagate through time O, H = self.rnn_layer_forward(X, H) # Backpropagate through time dW1, dW2, dW3, dbh, dbo, dh, loss = self.rnn_layer_backward(X, Y, H, O) #dW1, dW2, dW3, dbh, dbo = self.clip( dW1, dW2, dW3, dbh, dbo, maxValue=self.clipping_threshold) # Update parameters W1, dW2, dW3, dbh, dbo = self.update_parameters( dW1, dW2, dW3, dbh, dbo, learning_rate) return loss, dW1, dW2, dW3, dbh, dbo, H def gradient_checking(self, X, Y, epsilon=1e-5): # Forward propagate through time O, H = self.rnn_layer_forward(X, H=None) # Backpropagate through time dW1, dW2, dW3, dbh, dbo, dh, loss = self.rnn_layer_backward(X, Y, H, O) for param, dparam, name in zip([self.W1, self.W2, self.W3, self.bh, self.bo], [dW1, dW2, dW3, dbh, dbo], ['W1', 'W2', 'W3', 'bh', 'bo']): s0 = param.shape s1 = dparam.shape assert s0 == s1, 'Error! @ {} dimensions must match! and here {} != {} '.format(name, s0, s1) print('{}:'.format(name)) # number of checks for each parameter num_checks = 3 for i in range(num_checks): ri = int(np.random.uniform(0, param.size)) old_val = param.flat[ri] param.flat[ri] = old_val + epsilon # Forward propagate through time O, H = self.rnn_layer_forward(X, H=None) # Backpropagate through time dW1, dW2, dW3, dbh, dbo, dh, loss0 = self.rnn_layer_backward( X, Y, H, O) param.flat[ri] = old_val - epsilon # Forward propagate through time O, H = self.rnn_layer_forward(X, H=None) # Backpropagate through time dW1, dW2, dW3, dbh, dbo, dh, loss1 = self.rnn_layer_backward( X, Y, H, O) # restore the original value param.flat[ri] = old_val grad_analytical = dparam.flat[ri] grad_numerical = (loss0 - loss1) / (2 * epsilon) relative_error = abs( grad_analytical - grad_numerical) / abs(grad_numerical + grad_analytical) print('{}, {} => error: {} (error should be less than {})'.format( grad_analytical, grad_numerical, relative_error, 1e-7)) |
در پیاده سازی فوق ما مفاهیم پایه را در نظر گرفتیم . مفاهیمی همانند truncated backpropagation و مباحث مرتبط با مقداردهی اولیه و بحث محوشدگی گرادیان(روشهای مقابله با آن همانند Gradient clipping و…) بعد از بیان مفاهیم پایه بعدی در پستی جداگانه بیان میشوند.



[…] آموزش شبکه های عصبی بازگشتی (Recurrent neural network) بخش سوم : Backpr… […]
[…] آموزش شبکه های عصبی بازگشتی (Recurrent neural network) بخش سوم : Backpr… […]
e[np.arange(e.shape[0]), np.argmax(true_label, axis=1)] -=1
همانطور که توضیح داده اید خطا همان اختلاف خروجی شبکه در هر گام از مقدار واقعی همان گام است
اما من پیاده سازی که به صورت بالا گفته اید را متوجه نمیشوم
اگر امکان دارد این فرمول را بیشتر توضیح دهید
باتشکر
کدوم بخشش رو متوجه نمیشید؟
بخش مرتبط با پایتون (این دستور چکار میکنه) یا اینکه کلا مفهوم این قطعه کد رو متوجه نمیشید؟
اگر true_lable=[[1,0,0],[0,0,1]] حال np.argmax(true_label, axis=1)=[0,2]
و اگر خروجی شبکه به صورت e=[[0,1,0],[1,0 ,0]] (در واقع هنوز اموزش ندیده است) حال e.shape[0]=2 و [
[۰,۱]=np.arange(e.shape[0])
حال من متوجه نمیشوم
e[[0,1],[0,2]]یعنی چه ؟ و پس از آن e[[0,1],[0,2]]-=1چطور اختلاف مقادیر خروجی و واقعی را محاسبه میکند؟
سلام
کامنت های پیاده سازی نهایی که نوشتم رو بخونید متوجه میشید . این پیاده سازی vectorized هست . نمونه غیر برداری اون رو هم بصورت کامنت تو خط ۳۷ نوشتم که ببینید احتمالا کاملا متوجه میشید.
بخش اول سطرها رو مشخص میکنه و بخش دوم اندیس جاهایی که لیبل ۱ هست . مثلا سطر ۰ اندیس ۰ ( و سطر ۱ اندیس ۲ )
دقت بکنید میبینید سطر ۰ اندیس ۰ داره اطلاعات لیبل صحیح شما رو میده که برای نمونه اول (سطر اول) کدوم کلاس صحیح هست :
true_lable=[[1,0,0],
[۰,۰,۱]]
لیبل مربوط به نمونه اول (سطر ۰) اندیس ۰ نشانگر کلاس صحیح هست .
و در نمونه دوم (سطر ۱) هم اندیس ۲ نشانگر کلاس صحیح هست.
حالا همین درایه ها از ۱ کسر میشن تا خطا محاسبه بشه. (در هر گام زمانی ما فقط دنبال این هستیم که ببینیم در اون گام کلاس صحیح انتخاب شده یا نه و این رو از طریق قیاس خروجی پیش بینی شده – لیبل (که در اصل همون ۱ هست) بدست میاریم.
یعنی اینجوری که شما میفرمایید اگر من درست متوجه شده باشم
داخل ماتریس eفقط طبق مثال درایه های (۰,۰)و (۱,۲) منهی یک میشوند اگر مقدار این درایه ها برابر یک شده باشند پس با کم کردن از یک میشه صفر و این یعنی ما کاملا شبکمون اموزش دیده و به همون لیبل ها رسیده درسته؟ ودیگه مقادیر پارامترها اپدیت نمیشوند (چون مشتقات صفر خواهند شد ) .
و خطا per_ts_loss به چه علت استفاده شده است؟
ما داریم خطای خروجی رو محاسبه میکنیم برای یک گام! اگر جمله شما ۵ کلمه باشه باید مشخص بشه ایا تمام کلمات درست تشخیص داده شدند یا نه. پس شما هر بار یک کلمه رو میبینید آیا درست بوده تشخیص یا نه. صرف این که یک گام زمانی خروجی صحیحی تولید کرده به معنای تنظیم بودن شبکه نیست. انقدر این مساله تکرار میشه تا خطای کلی نهایتا کمینه بشه.
اون per_ts_loss هم برای محاسبه کراس انتروپی هست. کد رو ببینید مشخصه.
خیلی ممنون از توضیحاتتون